枝廣 龍哉 ≪遺伝統計学/呼吸器・免疫内科学≫ 日本人集団初の免疫細胞シングルセルアトラスの創生
〜多層オミクス情報をシングルセル空間へ投影するフレームワークを開発~
2025年7月28日
掲載誌 Nature Genetics
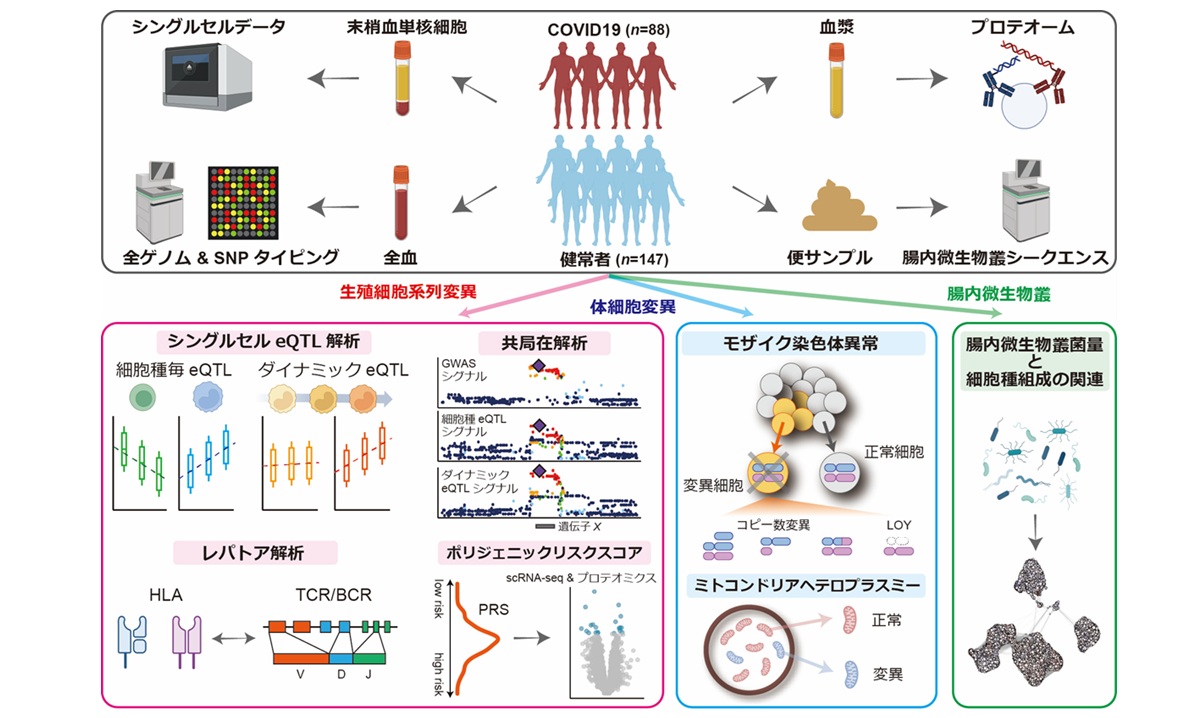
図1: 本研究の概要
クリックで拡大表示します
研究成果のポイント
- 日本人集団235名由来の150万超の末梢血単核細胞(PBMC) ※1を用いたシングルセルデータ※2を中心とし、ヒトゲノムデータ・血漿プロテオームデータ※3・腸内微生物叢シークエンスデータ※4の多層オミクス情報を有する免疫細胞シングルセルアトラス「OASIS(Osaka Atlas of Immune Cells)」を構築
- 各種オミクス情報をシングルセル空間に単一細胞解像度で投影することで、各オミクスが細胞種・細胞状態に応じて免疫細胞プロファイルを動的に制御していることを明らかに
- あわせて、日本人集団における初のシングルセルeQTL(expression quantitative trait loci)※5リソースの構築に成功し、日本人集団を対象とするゲノムワイド関連解析に活用できるデータベースに
- 加齢に伴い後天的に蓄積する体細胞変異※6が、COVID-19重症化に関与する分子メカニズムの一端を解明
- 新たな解析フレームワークが、疾患病態解明からゲノム創薬発展や個別化医療実現まで今後の広範な医学研究の強力な基盤となることに期待
概要
大阪大学大学院医学系研究科の枝廣龍哉助教(遺伝統計学/呼吸器・免疫内科学/理化学研究所生命医科学研究センター システム遺伝学チーム 客員研究員)、佐藤豪さん(当時:博士課程、現在:東京大学大学院医学系研究科 遺伝情報学 助教/理化学研究所生命医科学研究センター システム遺伝学チーム 客員研究員)、熊ノ郷淳教授(呼吸器・免疫内科学)、岡田随象教授(遺伝統計学/東京大学大学院医学系研究科 遺伝情報学 教授/理化学研究所生命医科学研究センター システム遺伝学チーム チームリーダー)らの研究グループは、日本人集団235名(新型コロナウイルス感染症:COVID-19患者88名、健常者147名)のPBMC 150万細胞超を対象にシングルセルトランスクリプトーム解析(scRNA-seq)※7を実施し、生殖細胞系列変異※8と体細胞変異を含むヒトゲノムデータ・血漿プロテオームデータ・腸内微生物叢シークエンスデータの多層オミクス情報を有する免疫細胞シングルセルアトラス「OASIS」を構築しました。
このOASISを用いて、各種オミクス情報をシングルセル空間に単一細胞解像度で投影する解析を網羅的に実施しました。その結果、生殖細胞系列変異(一塩基多型:SNP※9、HLA多型※10、ポリジェニックリスクスコア:PRS※11)、体細胞変異(モザイク染色体異常: mCA※12、Y染色体喪失: LOY※13、ミトコンドリア・ヘテロプラスミー※14)、腸内微生物叢菌量は、細胞種・細胞状態に応じて免疫細胞プロファイルを動的に制御していることが明らかとなりました。
また、その解析の一環で、日本人集団における初のシングルセルeQTLリソースを構築することに成功しました。
さらに、体細胞変異は、COVID-19患者において細胞種特異的に集約される傾向を示し、同一個体内においても変異細胞と正常細胞とで異なる免疫プロファイルを示すことが確認できました。また、これらの体細胞変異がCOVID-19重症化の一因となる分子メカニズムも解明しました。
本研究は、多層的オミクス情報をシングルセル空間に投影する新たな解析フレームワークを提示したものであり、今後の疾患病態解明やゲノム創薬発展に向けた重要な基盤となることが期待されます。
本研究の背景
近年のゲノムワイド関連解析(GWAS)※15により、多くの疾患や形質に関する数千もの感受性遺伝子領域が同定されてきました。しかし、GWASで同定されたこれらの遺伝子変異の大半は非コード領域※16に位置しており、その生物学的な機能を明らかにするのは困難であることが多いのが実情でした。こうした背景から、遺伝子変異が、遺伝子発現量・タンパク量などの分子形質に与える影響を調べるmolecular QTL※17解析が注目されるようになり、特にeQTL解析は、GWASで同定された遺伝子変異の機能的解釈において最も広く利用される解析手法として確立されています。
しかしながら、既存の大規模eQTLリソースの多くはバルクRNA-seqを用いたものであり、多様な細胞種や連続的な細胞状態に沿った動的な遺伝子発現制御機構など、全体像を評価することは困難でした。加えて、シングルセルeQTLリソースは欧米人集団を中心として構築されているため、日本人集団を対象としたシングルセルeQTLリソースは存在していませんでした。このことは、日本人集団におけるpost-GWAS解析※18において大きな障壁となっていました。
また、従来のeQTL研究において、再構成遺伝子であるT/B細胞受容体(TCR/BCR)※19の多様性に対する生殖細胞系列変異の影響を同時に検討したリソースは限定されていました。さらに、がん領域で注目されてきた体細胞変異を、非がんサンプル由来のPBMCにおいて“QTL”層として扱い、免疫細胞の性質や機能への影響をシングルセル解像度で解釈した先行研究はこれまで少ない状況でした。
本研究の内容
本研究グループは、日本人集団235名(COVID-19患者88名、健常者147名)由来の150万超のPBMCに対してシングルセル解析を実施し、生殖細胞系列変異と体細胞変異を含むヒトゲノムデータ・血漿プロテオームデータ・腸内微生物叢シークエンスデータの多層的オミクス情報が紐づいた、日本人集団初の免疫細胞シングルセルアトラス「OASIS(Osaka Atlas of Immune Cells)」を構築しました。
まず、このリソースを活用し、細胞種ごとの網羅的なシングルセルeQTL解析を実施、カタログ化するとともにその全シングルセルeQTL統計量の一般公開を行いました(https://japan-omics.jp/)。骨髄球系細胞※20クラスターにおいては、連続的な細胞状態に応じたeQTL(ダイナミックeQTL)解析を行い、細胞状態依存的な遺伝子発現制御機構を明らかにしました(図2)。また、東アジア人集団で実施されたGWASとOASISのシングルセルeQTLの共局在解析※21も実施し、疾患関連遺伝子多型がどの細胞種・細胞状態において、どの標的遺伝子の発現調節を介して表現型※22に関与しているかを定量的に評価しました。
さらに、HLA多型を含むゲノムワイドな生殖細胞系列変異とTCR/BCRレパトアの関連を解析・カタログ化しました。特にCOVID-19状況下のCD8陽性T細胞※23では、HLAクラスI遺伝子型※24により異なるTCRレパトアが誘導される可能性が示されました。COVID-19重症化PRSは、COVID-19状況下かつ細胞種特異的に、トランスクリプトームのみならずプロテオームにも影響を及ぼすことが明らかとなりました。腸内微生物叢菌量は、PBMCの細胞種組成に影響を与えていることが示され、これらの関連はシングルセル解像度の解析だからこそ初めて捉えることが可能となった現象でした。
体細胞変異に関しては、全ゲノムシーケンス※25とSNPタイピング※26の両方を用いることで、mCA、LOY、ミトコンドリア・ヘテロプラスミーを正確に定義しました。事前に同定した各サンプルが有する体細胞変異情報をシングルセル空間に投影することで、単一細胞解像度で変異細胞を同定、COVID-19状況下および細胞種特異的な変異の集約傾向の可視化に成功しました(図3)。さらに、同一個体内における変異細胞と正常細胞の比較から、mCAとLOYがCOVID-19重症化に寄与する分子メカニズムを明らかにしました。
 図2: ダイナミックeQTL解析の概要
図2: ダイナミックeQTL解析の概要
クリックで拡大表示します
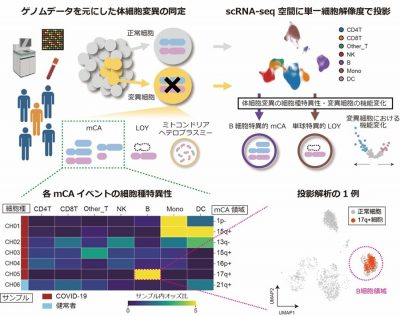 図3: 体細胞変異の同定とシングルセル空間への投影解析の概要
図3: 体細胞変異の同定とシングルセル空間への投影解析の概要
クリックで拡大表示します
本研究が社会に与える影響(本研究成果の意義)
本研究は、日本人集団初の免疫細胞シングルセルアトラス「OASIS」の構築のみならず、様々なヒトゲノム変異・プロテオーム・メタゲノムといった多層オミクス情報をシングルセル空間に投影する新たな解析フレームワークを提示した点で、疾患病態解明から個別化医療の実現に至る広範な展開が期待される、非常に先進的な成果と考えられます。
GWASで同定された疾患関連遺伝子変異のより高解像度な機能的解釈、免疫細胞の個人内・個体間多様性の理解、さらにはウイルス感染症や自己免疫疾患などの病態解明においても、本リソースと開発された解析手法は、今後の医学研究の強力な基盤となることが期待されます。
研究者コメント
<枝廣 龍哉 助教>
私たちは本研究を通じて、日本人集団における免疫細胞の多様性とその分子基盤を、シングルセル解析と多層オミクス情報の統合により詳細に描き出すことに成功しました。特に、COVID-19の重症化に関わる体細胞変異の意義や、疾患関連遺伝子の細胞種特異的な作用メカニズムに迫ることができたのは、本プロジェクトがもつリソースの強みの賜物です。この成果が、今後の自己免疫疾患や感染症、さらには個別化医療やゲノム創薬の研究に広く活用されることを期待しています。
本研究は、大阪大学を中心に、多分野・多機関にまたがる共同研究体制によって支えられてきました。ご協力いただいた全ての共同研究者、技術支援スタッフ、研究資金提供機関、そして貴重な検体をご提供くださった皆様に、心より御礼申し上げます。
用語説明
※1 末梢血単核細胞(PBMC)
血液中から単離される単核細胞群で、主にリンパ球や単球などの免疫細胞で構成される。免疫応答や疾患研究の重要な解析対象。
※2 シングルセルデータ
個々の細胞ごとに得られた分子情報(例:遺伝子発現量や受容体配列など)を指し、細胞集団内の多様性や状態変化を単一細胞解像度で解析可能とする。
※3 血漿プロテオームデータ
血漿中に含まれるタンパク質全体を網羅的に解析したデータ。疾患の診断・予後予測やバイオマーカー探索に有用。
※4 腸内微生物叢シークエンスデータ
腸内に存在する細菌の種類や量を網羅的に明らかにするためのメタゲノム解析データ。宿主の免疫や代謝への影響が注目されている。
※5 eQTL(expression quantitative trait loci)
遺伝子発現量の個人差と関連するゲノム領域。このような遺伝子発現量に対する遺伝子多型の影響をeQTL効果と呼ぶ。
※6 体細胞変異
生涯のうちに後天的に獲得される遺伝子変異で、生殖細胞には伝わらない。加齢や環境要因により蓄積し、疾患リスクに関与することがある。
※7 シングルセルトランスクリプトーム解析(scRNA-seq)
1細胞ごとのmRNAを網羅的に解析し、各細胞の遺伝子発現プロファイルを高解像度で取得できる次世代型の解析技術。
※8 生殖細胞系列変異
親から子へ遺伝する変異で、生涯を通じて全身の細胞に共通して存在する。疾患感受性や体質の個人差に関与する。
※9 一塩基多型(SNP)
ヒトゲノムの特定の位置における1塩基の違いで、個人間の遺伝的多様性を構成する最も一般的な変異形式。
※10 HLA多型
ヒト白血球型抗原(HLA)の遺伝子領域に見られる多様性。免疫応答や感染症、自己免疫疾患などとの関連が知られる。
※11 ポリジェニックリスクスコア(PRS)
ヒトゲノム配列上に存在する遺伝子変異のうち、疾患との関連が示唆された数十〜数十万の遺伝子変異について、効果量の重み付きの和を個人ごとに計算したスコア。このスコアは実際の疾患発症リスクと相関することが示されており、スコアを計算することで疾患にかかりやすい遺伝的体質を持っているかどうか調べることができる。
※12 モザイク染色体異常(mCA)
mosaic chromosomal alteration。一つの個体内に、遺伝的に異なる細胞集団が共存する状態。主に体細胞で生じる染色体構造の変化が原因で、加齢や疾患との関連が報告されている。
※13 Y染色体喪失(LOY)
男性に特有のY染色体が一部の細胞で後天的に失われる現象。加齢とともに増加し、疾患との関連も示唆されている。
※14 ミトコンドリア・ヘテロプラスミー
一つの細胞内に複数種類のミトコンドリアDNAが共存している状態。加齢や病態によって頻度が変動する。
※15 ゲノムワイド関連解析(GWAS)
Genome Wide Association Study。ヒトゲノム配列上に存在する数百万~数千万か所の遺伝子変異と疾患や形質との発症の関係を網羅的に検討する、遺伝統計解析手法。
※16 非コード領域
タンパク質をコードしないゲノム領域。近年、転写調節やエピジェネティクスなどの機能的役割が注目されている。
※17 molecular QTL
遺伝子変異が分子レベルの形質(例:遺伝子発現量、タンパク質量など)に与える影響を調べる解析。遺伝子変異の生物学的機能を調べる際に有用であり、molecular QTL解析でよく用いられる。
※18 post-GWAS解析
ゲノムワイド関連解析(GWAS)によって同定された疾患関連遺伝子多型の生物学的機能や病態への関与を解明するための後続解析の総称。molecular QTL解析や共局在解析などを通じて、疾患関連多型がどの遺伝子の発現や機能に影響し、どの細胞種・状態で作用するかを明らかにすることを目的とする。GWASで得られた統計的な関連を、病態メカニズムの理解や創薬応用につなげる上で不可欠な解析ステップである。
※19 T/B細胞受容体(TCR/BCR)
T細胞およびB細胞が抗原を認識するために持つ受容体。個々の免疫細胞が固有のレパトアを持ち、適応免疫の中核を担う。
※20 骨髄球系細胞
造血幹細胞から分化する免疫細胞の一群で、単球、好中球、好酸球、好塩基球、樹状細胞などを含む。主に自然免疫に関与する。本研究においては、単球と樹状細胞が該当する。
※21 共局在解析
遺伝子多型と疾患などの表現系との関連を調べるGWASシグナルと、遺伝子多型が分子形質との関連を調べるmolecular-QTLシグナルが、同一ゲノム領域において、オーバーラップしているか(=共局在)を統計的に検出する解析手法。
※22 表現型
遺伝情報と環境要因の相互作用によって現れる個体の観察可能な特徴(例:疾患の有無、身長、免疫応答など)を指す。
※23 CD8陽性T細胞
T細胞の一種で、主にウイルス感染細胞や腫瘍細胞の排除に関与する。HLAクラスI分子を介して抗原を認識する。
※24 HLAクラスI遺伝子型
HLA遺伝子群の中でもクラスIに属するHLA-A、HLA-B、HLA-Cなどの遺伝子の型(アリル)を指す。主にCD8陽性T細胞による抗原認識に関与する。
※25 全ゲノムシーケンス
個人の全DNA配列を網羅的に読み取る解析手法。遺伝子変異や構造変化を高精度で検出可能であり、包括的なゲノム情報の取得に用いられる。
※26 SNPタイピング
特定の一塩基多型(SNP)の有無や型を調べる遺伝子解析技術。高スループットに多数の個体を対象とした遺伝子型決定が可能で、GWASなどに広く用いられる。
特記事項
本研究成果は、2025年7月28日(月)18時(日本時間)に英国科学誌「Nature Genetics」(オンライン)に掲載されました。
【タイトル】
“Deciphering state-dependent immune features from multi-layer omics data at single-cell resolution”
【著者名】
Ryuya Edahiro1-3,#†, Go Sato1,3,4,#, Tatsuhiko Naito1,3, Yuya Shirai1,2,5, Ryunosuke Saiki6, Kyuto Sonehara1,3,7, Yoshihiko Tomofuji1,3,7, Kenichi Yamamoto1,8,9, Shinichi Namba1,3,7, Noah Sasa1,3,10, Genta Nagao7,11, Qingbo S Wang1,7, Yugo Takahashi1, Takanori Hasegawa12, Toshihiro Kishikawa1,10,13, Ken Suzuki1,14, Yu-Chen Liu15, Daisuke Motooka15-17, Ayako Takuwa15, Hiromu Tanaka11, Shuhei Azekawa11, Japan COVID-19 Task Force*, Ho Namkoong18, Ryuji Koike19, Akinori Kimura20, Seiya Imoto21, Satoru Miyano12, Takanori Kanai22, Koichi Fukunaga11, Mamoru Uemura4, Takayoshi Morita2,23, Yasuhiro Kato2,23, Haruhiko Hirata2, Yoshito Takeda2, Yuichiro Doki4, Hidetoshi Eguchi4, Daisuke Okuzaki15-17,24,25, Shuhei Sakakibara26,27, Seishi Ogawa6,28, Atsushi Kumanogoh2,17,23-25,29,† and Yukinori Okada1,3,5,7,30,†
- 大阪大学大学院医学系研究科 遺伝統計学
- 大阪大学大学院医学系研究科 呼吸器・免疫内科学
- 理化学研究所 生命医科学研究センター システム遺伝学チーム
- 大阪大学大学院医学系研究科 消化器外科学
- 大阪大学免疫学フロンティア研究センター(IFReC) 免疫統計学
- 京都大学大学院医学研究科 腫瘍生物学
- 東京大学大学院医学系研究科 遺伝情報学
- 大阪大学大学院医学系研究科 小児科学
- 大阪大学大学院医学系研究科 保健学専攻
- 大阪大学大学院医学系研究科 耳鼻咽喉科・頭頸部外科
- 慶應義塾大学医学部 内科学教室(呼吸器)
- 東京医科歯科大学 M&Dデータ科学センター
- 愛知県がんセンター病院 頭頸部外科
- 東京大学大学院医学系研究科 糖尿病・代謝内科
- 大阪大学免疫学フロンティア研究センター(IFReC) ヒト免疫学(シングルセルゲノミクス)
- 大阪大学微生物病研究所 ゲノム情報解析センター
- 大阪大学先導的学際研究機構(OTRI) 生命医科学融合フロンティア研究部門
- 慶應義塾大学医学部 感染症学
- 東京医科歯科大学 健康医科学イノベーション開発研究センター(HeRD)
- 東京医科歯科大学 研究推進機構
- 東京大学医科学研究所 ヒトゲノム解析センター 医療数理情報学分野
- 慶應義塾大学医学部 内科学教室(消化器)
- 大阪大学免疫学フロンティア研究センター(IFReC) 感染病態
- 大阪大学感染症総合教育研究拠点(CiDER)
- 日本医療研究開発機構(AMED) 革新的先端研究開発支援事業
- 大阪大学免疫学フロンティア研究センター(IFReC) 免疫統計学
- 慈慶大学保健医療学部 医療安全管理学研究科
- 京都大学 ヒト生物学高等研究拠点(WPI-ASHBi)
- 大阪大学ワクチン開発拠点 先端モダリティ・DDS研究センター(CAMaD)
- 大阪大学 ヒューマン・メタバース疾患研究拠点(WPI-PRIMe)
(†責任著者、#同等貢献、*コロナ制圧タスクフォース研究グループ全員のリストは論文中に記載)
DOI:10.1038/s41588-025-02266-3
本研究は、国立研究開発法人日本医療研究開発機構(AMED)、日本医療研究開発機構–戦略的創造研究推進事業(AMED-CREST)事業、JSPS科研費、国立研究開発法人科学技術振興機構(JST)ムーンショット型研究開発事業、武田科学振興財団、大阪大学大学院医学系研究科バイオインフォマティクス・イニシアティブ、大阪大学先導的学際研究機構(OTRI)、大阪大学感染症総合教育研究拠点(CiDER)、AMED SCARDAワクチン開発のための世界トップレベル研究開発拠点の形成事業「ワクチン開発のための世界トップレベル研究開発拠点群大阪府シナジーキャンパス(大阪大学ワクチン開発拠点)」(JP223fa627002)、理化学研究所科学研究基盤モデル開発プログラム(TRIP-AGIS)、小野薬品がん・免疫・神経研究財団、日本応用酵素協会の支援を受けて行われました。
本件に関して、オンラインにて記者発表を行いました。











