友藤 嘉彦、岡田 随象 ≪遺伝統計学≫ 日本人集団の腸内細菌叢・ウイルス叢の特徴を解明
~データベース構築で食生活や病気との関わりを明らかに~
2022年12月 1日
掲載誌 Cell Genomics
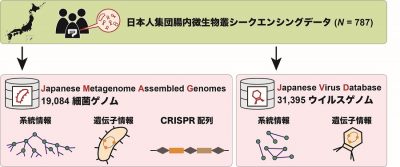 図1. データベース構築の概要
図1. データベース構築の概要
クリックで拡大表示します
研究成果のポイント
- 日本人集団787名の腸内微生物叢※1シークエンス情報から、細菌・ウイルスのゲノム配列を再構築し、データベース化した
- 日本人集団の腸内細菌データベース(JMAG: Japanese Metagenome Assembled Genomes)には、納豆や海苔などの日本食に関連する細菌・遺伝子のゲノム配列が含まれていた
- 日本人集団の腸内ウイルスデータベース(JVD: Japanese Virus Database)と複数データベースとの統合解析によって、腸内ウイルスと病気・人種集団との関連を明らかにした
概要
大阪大学大学院医学系研究科の友藤嘉彦さん(博士課程)(遺伝統計学)、岡田随象 教授(遺伝統計学/理化学研究所生命医科学研究センター システム遺伝学チーム チームリーダー/東京大学大学院医学系研究科 遺伝情報学 教授)らの研究グループは、日本人集団787名の腸内微生物叢シークエンス情報※2から、細菌・ウイルスのゲノム配列を再構築し、データベース化しました(図1)。
また、今回開発したデータベースと、既存の開学データベースとの統合解析により、以下のような日本人集団の腸内細菌の特徴、及び海外を含めた食事・病気・人種集団と腸内微生物との関係を明らかにしました。
・構築した腸内細菌ゲノムデータベース中には日本食(海苔・納豆)に関連した細菌・遺伝子情報が含まれていました。
・構築した腸内ウイルスゲノムデータベースを用いて、crAss-like phage※3に着目した解析を行ったところ、β crAss-like phageがアジアや欧米の人で少ない一方、アフリカやオセアニアの人で多いことが分かりました。
・病気とcrAss-like phageとの関連を評価したところ、複数亜科のcrAss-like phageが関節リウマチ※4、全身性エリテマトーデス※5、炎症性腸疾患※6において減少していることがわかりました。
本研究成果によって、食事・病気・人種集団と腸内微生物との関係の一端が明らかになったとともに、大規模な日本人集団の腸内微生物ゲノムデータベースが構築されました。構築された微生物ゲノムデータベースは、今後の医学・生物学研究に資する重要な研究資源になると期待されます
研究の背景
我々の腸内には、細菌やウイルスなど、数多くの微生物が存在し、腸内微生物叢を構成しています。腸内微生物叢は免疫反応や代謝応答を介して我々の体に大きな影響を与えており、多くの病気との関連が既に示されています。それゆえに、腸内微生物叢を対象とした医学・生物学研究がこれまでに数多く行われてきました。腸内微生物研究において、個々の微生物のゲノム情報は必要不可欠な研究資源です。古くから、微生物を単離・培養し、そのゲノムをシークエンスすることで、個々の微生物のゲノム情報を得てきました。しかし、この方法には、手間や時間がかかる上に、培養が難しい細菌やウイルスなどに適用しにくいという問題がありました。そこで、近年、単離・培養を行わずに、腸内微生物叢全体のシークエンス情報から、個々の微生物のゲノム情報(metagenome assembled genome: MAG※7)が再構築されるようになってきました。
世界各国で、細菌ゲノムの再構築・カタログ化が行われ、既存のデータベースには多種多様な腸内細菌ゲノムが登録されるようになりました。一方で、多くの既存データベースに含まれる細菌ゲノムの大半は欧米人集団や中国人集団に由来するものであり、日本人集団に由来するものは殆ど含まれていませんでした。また、先行研究の多くは、腸内細菌のみを対象としており、腸内ウイルス、特に細菌に感染するウイルスであるバクテリオファージのゲノム情報については、殆ど含まれていませんでした。
本研究の成果
今回、研究グループでは、日本人集団787名の腸内微生物叢シークエンス情報に対して、独自に開発した腸内微生物ゲノム再構築パイプラインを適用し、19,084の細菌ゲノムと31,395のウイルスゲノムをそれぞれJapanese Metagenome Assembled Genomes(JMAG)とJapanese Virus Database(JVD)としてデータベース化しました。そして、JMAGとJVDを用いて、腸内微生物と食事・病気・人種集団との関連を評価しました。
研究チームは、JMAGと海外の細菌ゲノムデータベースとの比較解析を行い、日本人集団の腸内に特異的な細菌・遺伝子を探索しました。その結果、JMAG中には納豆菌※8のゲノムが多く含まれていました。また、海苔に含まれる炭水化物を分解する酵素であるβ-porphyranase※9もJMAG中に特に多く含まれていました。つまり、JMAG中には日本食に関連した細菌・遺伝子が多く含まれていました。
日本人集団の食生活には、ヒトゲノムの多様性、特にお酒を飲めるかどうかを規定する遺伝子多型としても知られるrs671※10が関連することが知られています。岡田教授らは、rs671と乳製品消費量との関連を過去に報告していました。今回の研究では、rs671と乳製品に関連する腸内細菌2種(Enterococcus_B, Streptococcus thermophilus)との関連が明らかになりました。このことから、ヒトゲノムの多様性が食生活への影響を介して、腸内細菌に影響を与えている可能性が示唆されました。
研究チームは、JVDと複数の既存データベースとを統合し、ウイルスの系統分類を行いました。その結果、JVDに含まれる種のうち半分以上は既存データベースに含まれていない種であることがわかりました。次に、研究チームは、ウイルスゲノムの中でも、特にcrAss-like phageというバクテリオファージに着目しました。crAss-like phageは2014年と比較的最近に発見されたウイルスの科で、ヒトの腸内に多く存在することから、今世界中で着目されています。研究チームは、crAss-like phageを亜科レベルまで分類した上で、地域ごとの腸内における組成の違いを調べました。その結果、β crAss-like phageは日本をはじめとしたアジア諸国や欧米諸国では少ない一方で、アフリカ諸国やオセアニア諸国では多いということがわかりました。さらに、crAss-like phageと様々な病気との関連を調べたところ、関節リウマチ、全身性エリテマトーデス、炎症性腸疾患では複数亜科のcrAss-like phageが減少していることがわかりました。興味深いことに、crAss-like phageは、腸内細菌の多様性と正の相関を持っていました。腸内細菌の多様性の低下は、腸内細菌叢の破綻(ディスバイオシス)においてみられる主要な変化の一つです。このことから、crAss-like phageが腸内細菌叢の健康さの指標になりうる可能性が示唆されました。
最後に、研究チームはCRISPR※11を利用した解析によって、JVD中のウイルスがJMAG中のどの細菌に感染しているのかを網羅的に探索しました。その結果、crAss-like phageがBacteroidota門やFirmicutes門に属する様々な細菌に感染していることがわかりました(図3)。
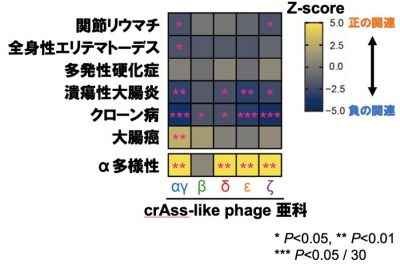 図2. crAss-like phageと病気との関連解析
図2. crAss-like phageと病気との関連解析
ヒートマップの色は病気及びα多様性とcrAss-like phageとの関連の強さを表す
ヒートマップ中のアスタリスクは各関連が有意水準を満たしているかどうかを表す
クリックで拡大表示します
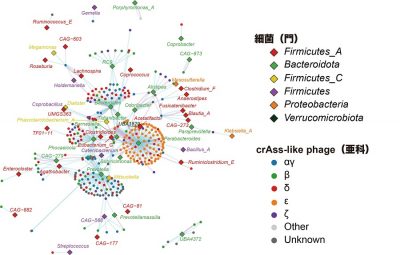 図3. crAss-like phageと細菌の感染–被感染ネットワーク
図3. crAss-like phageと細菌の感染–被感染ネットワーク
個々の菱形の点は細菌の属を、個々の丸点はcrAss-like phageの種を表す
菱形の点の色は細菌の門を、丸点の色はcrAss-like phageの亜科を指す
点同士を結ぶ線は感染–被感染関係を表す
クリックで拡大表示します
本研究が社会に与える影響(本研究成果の意義)
本研究成果によって、食事・病気・人種集団と腸内微生物叢との関連が新たに見出されました。本研究で食事・病気・人種集団との関連が見出された微生物については、今後、動物実験などのさらなる検証を進めることで、病気の治療標的やプロバイオティクスとしての利用が可能になると期待されます。
また、本研究で構築された微生物ゲノムデータベース(JMAG・JVD)は公開されており、研究者が自由に利用することができます。JMAGやJVDは、日本人集団の腸内細菌・ウイルス叢の多様性を考慮した解析を実施可能にすると考えられ、今後、腸内微生物研究を行なっていく上で、重要な研究資源になっていくと期待されております。
研究者のコメント
<友藤 嘉彦 大学院生>
本研究では日本人集団の腸内微生物ゲノムデータベースを再構築することに成功しました。我々の構築したデータベースが、世界中の多くの研究者たちに利用され、医学・生物学研究の今後の発展に貢献できることを、心より願っております。本研究は大阪大学医学部附属病院や関連施設より提供していただいたサンプルを用いることで達成することができました。全ての共同研究者や研究支援機構、並びにサンプルを提供してくださった方々に深く感謝を申し上げます。
用語説明
※1 腸内微生物叢
宿主であるヒトや動物と共生関係にある多種多様な腸内微生物の集まり。
※2 腸内微生物叢シークエンス
微生物の全ゲノムDNAを短いDNA鎖に切断してライブラリを作成し、次世代シークエンサーによって配列決定する手法。
※3 crAss-like phage
バクテリオファージの分類の一つで科レベル相当と考えられている。2014年にヒトの腸内細菌叢メタゲノムデータから再構築され、発見された。ヒトの腸内における主要なバクテリオファージの1つ。
※4 関節リウマチ
自己免疫疾患の1つで、関節に炎症が起き、骨が破壊されて、放っておくと関節が変形してしまう病気。
※5 全身性エリテマトーデス
自己免疫疾患の1つで、自己抗体の産生や、腎臓・皮膚・脳神経・心臓などに起きる多様な臓器障害を特徴とする病気。
※6 炎症性腸疾患
自己免疫疾患の1つで、自分の免疫細胞が腸の細胞を攻撃してしまうことで腸に炎症を起こす病気。潰瘍性大腸炎やクローン病を含む。
※7 metagenome assembled genome:MAG
腸内微生物叢シークエンス情報から、再構築した微生物由来ゲノムの総称。腸内微生物叢シークエンシングによって得られた短いゲノム配列情報をつなぎ合わせたり、グループ化したりすることによって、個々の微生物のゲノムを再構築することができる。
※8 納豆菌
納豆の製造に必要な細菌。枯草菌の一種で学名はBacillus subtilis var. natto。
※9 β-porphyranase
紅藻(アマノリ属の一部で食用の海苔の原料の1つ)に含まれる硫酸化炭水化物であるポルフィランを分解する酵素。
※10 rs671
アルコールの代謝に関わる遺伝子(ALDH2)中に存在する遺伝子多型、つまり個人間でのゲノム配列の違い。東アジア人集団に特異的に存在することが知られている。
※11 CRISPR
原核生物が持つファージやプラスミドに対する獲得免疫機構の1つ。侵入してきたファージやプラスミドのDNA配列の一部をスペーサーとしてCRISPR領域に取り組み、免疫記憶として利用する。本研究においては、CRISPR領域中のスペーサー配列とウイルス配列との相同性を調べることにより、細菌がどの様なウイルスに感染したのかを見出した。
特記事項
本研究成果は、2022年12月1日(木)午前1時(日本時間)に英国科学誌「Cell Genomics」(オンライン)で掲載されました。
【タイトル】
“Prokaryotic and viral genomes recovered from 787 Japanese gut metagenomes revealed microbial features linked to diets, populations, and diseases”
【著者名】
Yoshihiko Tomofuji1,2*, Toshihiro Kishikawa1,3,4, Yuichi Maeda2,5,6, Kotaro Ogawa7, Yuriko Otake-Kasamoto8, Shuhei Kawabata9, Takuro Nii5,6, Tatsusada Okuno7, Eri Oguro-Igashira5,6, Makoto Kinoshita7, Masatoshi Takagaki9, Naoki Oyama10, Kenichi Todo7, Kenichi Yamamoto1,11,12, Kyuto Sonehara1,2, Mayu Yagita5,6, Akiko Hosokawa13, Daisuke Motooka2,14, Yuki Matsumoto14, Hidetoshi Matsuoka15, Maiko Yoshimura15, Shiro Ohshima15, Shinichiro Shinzaki8, Shota Nakamura2,14, Hideki Iijima8, Hidenori Inohara3, Haruhiko Kishima9, Tetsuo Takehara8, Hideki Mochizuki7, Kiyoshi Takeda6,16, Atsushi Kumanogoh2,5,17, Yukinori Okada1,2,12,18,19,20* (*責任著者)
【所属】
- 大阪大学大学院医学系研究科 遺伝統計学
- 大阪大学先導的学際研究機構(OTRI) 生命医科学融合フロンティア研究部門
- 大阪大学大学院医学系研究科 耳鼻咽喉科・頭頸部外科学
- 愛知県がんセンター 頭頸部外科部
- 大阪大学大学院医学系研究科 呼吸器・免疫内科学
- 大阪大学大学院医学系研究科 免疫制御学
- 大阪大学大学院医学系研究科 神経内科学
- 大阪大学大学院医学系研究科 消化器内科学
- 大阪大学大学院医学系研究科 脳神経外科学
- 川崎医科大学 脳卒中医学
- 大阪大学大学院医学系研究科 小児科学
- 大阪大学免疫学フロンティア研究センター(IFReC) 免疫統計学
- 吹田市民病院 脳神経内科
- 大阪大学微生物病研究所(RIMD) 感染症メタゲノム研究分野
- 大阪南医療センター リウマチ・膠原病・ アレルギー科
- 大阪大学免疫学フロンティア研究センター(IFReC) 粘膜免疫学
- 大阪大学免疫学フロンティア研究センター(IFReC) 感染病態分野
- 理化学研究所生命医科学研究センター システム遺伝学チーム
- 大阪大学感染症総合教育研究拠点(CiDER)
- 東京大学大学院医学系研究科 遺伝情報学
【DOI番号】10.1016/j.xgen.2022.100219
本研究は、国立研究開発法人日本医療研究開発機構(AMED)革新的先端研究開発支援事業FORCE「メタゲノムワイド関連解析による疾患特異的微生物叢解明と個別化医療実装」及びゲノム医療実現推進プラットフォーム事業・先端ゲノム研究開発(GRIFIN)「遺伝統計学に基づく日本人集団のゲノム個別化医療の実装」の一環として行われ、大阪大学免疫学フロンティア研究センター次世代主任研究者支援プログラム、大阪大学先導的学際研究機構、大阪大学大学院医学系研究科バイオインフォマティクスイニシアティブの協力を得て行われました。










